Das Keimspektrum des Endodonts
Dank der besseren technischen Möglichkeiten für die Therapie endodontischer Infektionen – zum Beispiel durch den Einsatz von OP-Mikroskopen, maschinellen Aufbereitungsmethoden, der Einführung definierter Spülprotokolle und verbesserter Obturationsmethoden – nahmen auch die Erfolgsquoten der Behandlungen zu. Dennoch gibt es nach wie vor – manchmal für den Zahnarzt unerklärliche – Misserfolge (Abbildung 1). Neben physikalischen Ursachen wie beispielsweise Wurzellängsfrakturen sind dafür in erster Linie Mikroorganismen verantwortlich.
Mit neuen Analysemethoden und einem neuen Verständnis der bakteriellen Besiedelung der Wurzelkanäle lassen sich möglicherweise Therapien entwickeln, mit denen sich auch diese „Problemzähne“ erhalten lassen. Aussagen zur mikrobiellen Besiedelung infizierter Wurzelkanäle und zur Wirksamkeit von neuen Therapieoptionen lassen sich allerdings nur überprüfen, wenn man den wissenschaftlichen Hintergrund versteht.
Die Hauptursache für Misserfolge: Infektionen
Bereits vor gut einem halben Jahrhundert wurden Mikroorganismen als Ursache für die Inflammation (apikale Parodontitis) an der Kontaktstelle zwischen dem infizierten Wurzelkanal und dem umgebenden Gewebe (apikale oder laterale Foramina) nachgewiesen [Kakehashi et al., 1965]. Wurzelkanalinfektionen werden primär durch eine mikrobielle Kolonisation des Wurzelkanals durch Mikroorganismen verursacht, während eine sekundäre endodontische Infektion nach einer regulären Wurzelkanalbehandlung erfolgt [Siqueira und Rôças, 2009; Kirkevang et al., 2007; Moreno et al., 2013].
Sekundäre endodontische Infektionen können durch die persistierende Kontamination mit Mikroorganismen oder durch die Reinfektion aufgrund einer nicht adäquaten Behandlung der Wurzelkanäle beziehungsweise durch eine nicht suffiziente postendodontische Restauration verursacht werden [Schirrmeister et al., 2007]. Auch wenn eine hundertprozentige Eradikation der Mikroorganismen in den infizierten Wurzelkanälen nicht möglich ist, kann eine starke Reduktion der Infektionsmikrobiota zur Verringerung der Pathogenität der unspezifischen endodontischen Infektion führen. Die Beurteilung der ökologischen Bilanz endodontischer Infektionen setzt die genaue Kenntnis der beteiligten Mikroorganismen voraus.
Studien zufolge wurden aus 44 bis 100 Prozent der Wurzelkanäle mit endodontischen Behandlungsfehlern und notwendiger Revision der Wurzelfüllungen Mikroorganismen isoliert [Iriboz et al., 2020, Schirrmeister et al., 2009]. Aufgrund der Exposition der Zähne zu einer hohen Diversität oraler Mikroorganismen ist die endodontische Infektion in der Regel unspezifisch, so dass verschiedene Spezies beteiligt sind.
Das orale Mikrobiom mit über 700 verschiedenen Arten, die den Phyla Actinobacteria, Bacteroidetes, Chlamydiae, Chloroflexi, Euryarchaeota, Firmicutes, Fusobacteria, Proteobacteria, Spirochaetes, SR1, Synergistetes, Tenericutes und TM7 zuzuordnen sind, ist das zweitgrößte humane Mikrobiom nach der Darmflora [Dewhirst et al., 2010; Wade, 2013; Zehnder und Belibsakis, 2015]. Daher ist es plausibel, dass die Kenntnis der mikrobiellen Diversität endodontischer Infektionen für das Verständnis der Wechselwirkung mit dem Immunsystem des Wirts sowie für die Entwicklung neuer Wurzelkanal-Behandlungsmethoden unerlässlich ist [Tzanetakis et al., 2015; Siqueira und Rôças, 2009].
Da das Mikrobiom des Wurzelkanals in Form von Biofilm-ähnlichen Strukturen mit einem Anteil an anaeroben Keimen vorliegt [Ricucci und Siqueira, 2010], ist eine vollständige Identifizierung der endodontischen Infektionsflora mittels Kulturtechnik nicht möglich. In diesem Zusammenhang haben Zehnder und Belibasakis in einem Review über die Dynamik von Wurzelkanalinfektionen speziell auf die Problematik der Kultivierung anaerober Bakterien in den unterschiedlichen Zonen der Wurzelkanäle hingewiesen [Zehnder und Belibasakis, 2015]. Erst mit der Anwendung der kulturunabhängigen 16S-rDNA-PCR-basierten Techniken zum Nachweis nichtkultivierbarer Wurzelkanal-Bakterien – wie der Einsatz speziesspezifischer Primer, Klonierung und Sequenzierung der 16S rDNA-Gene, Denaturierungs-Gradienten-Gel-Elektrophorese (denaturing gradient gel electrophoresis, DGGE), terminaler Restriktionsfragment-Längen-Polymorphismus (terminal restriction fragment length polymorphism, T-RFLP) – konnte gezeigt werden, dass die Diversität der endodontischen Mikroflora wesentlich größer ist, als es durch die klassische Kulturtechnik bekannt war [Siqueira und Rôças, 2005; Siqueira et al., 2004; Saito et al., 2006, 2010; Anderson et al., 2013]. Mittels der 16S-rDNA-PCR und der Klonierungstechnik haben Paster et al. bereits 2001 nachgewiesen, dass bis zu 50 Prozent der oralen Bakterien mittels Kulturtechnik nicht nachweisbar sind.
Diese Erkenntnisse führen zu einem Paradigmenwechsel in der Beschreibung der Ätiologie und möglicherweise zukünftig auch in der Therapie von refraktären periapikalen Entzündungen. Wurde in den vergangenen Jahren vielfach die Persistenz von Enterococcus faecalis als isolierter Keim für die Misserfolge von endodontischen Behandlungen verantwortlich gemacht, so konnte mithilfe von kulturunabhängigen Techniken (insbesondere der Klonierungstechnik) gezeigt werden, dass die Rolle von Enterococcus faecalis für endodontische Infektionen möglicherweise überschätzt wurde [Sakamoto et al., 2008; Anderson et al., 2013].
Die Klonierungstechnik hat den Nachweis von mehr Arten ermöglicht, die mittels Kulturtechnik nicht isolierbar waren und trug damit zu einer markanten Erweiterung des Verständnisses endodontischer Infektionen bei [Saito et al., 2006; Sakamoto et al., 2008; Anderson et al., 2013]. Die Klonierungstechnik hat jedoch auch Methoden-immanente Limitationen, da sie hochfrequente Mikroorganismen bevorzugt nachweist [Özok et al., 2012].
Neben den Einschränkungen, die durch die mikrobiologischen Analysemethoden verursacht werden, spielt die Probenentnahme insbesondere für endodontische Infektionen eine kritische Rolle. Der komplizierte Aufbau der Wurzelkanäle macht es unmöglich, die tatsächlich vorhandene Infektionsflora aus allen Nischen (Seitenkanäle, Pulpaperiodontalkanäle, apikales Delta) zu entnehmen [Nair et al., 2005; Sathorn et al., 2007].
Prof. Dr. Ali Al-Ahmad

1985–1986: Besuch des Studienkollegs an der Universität Heidelberg
1986–1993: Biologiestudium an der Fakultät für Biologie der Albert-Ludwigs-Universität Freiburg mit dem Abschluss „Diplom“
1994–1999: Wissenschaftlicher Angestellter des Instituts für Umweltmedizin und Krankenhaushygiene am Universitätsklinikum Freiburg
1997–1999: Stellvertretender Ressortleiter der Abteilung Umweltforschung am Institut für Umweltmedizin und Krankenhaushygiene in Freiburg
1998: Abschluss der Dissertation am Institut für Umweltmedizin und Krankenhaushygiene
1998–1999: Postdoc am Institut für Umweltmedizin und Krankenhaushygiene
2000–2004: Wissenschaftlicher Mitarbeiter und S2-Laborleiter der GeneScan Europe AG
seit 2004: Wissenschaftlicher Mitarbeiter an der Klinik für Zahnerhaltungskunde und Parodontologie
seit 2006: Laborleiter und Leiter des Bereichs „Orale Mikrobiologie“ an der Klinik für Zahnerhaltungskunde und Parodontologie
2011: Habilitation in der experimentellen Zahnmedizin an der Klinik für Zahnerhaltungskunde und Parodontologie des Universitätsklinikums Freiburg (Mentor: Prof. Dr. Elmar Hellwig)
2014: Verleihung des Titels Apl-Professor
Bei sekundären Infektionen ist auch das Wurzelkanalfüllungsmaterial bakteriell kontaminiert. Hier ist klinisch eine extrem sorgfältige Entfernung für einen Behandlungserfolg erforderlich. Will man nun untersuchen, welche Keime in diesen Wurzelkanälen vorhanden sind, muss man auch das Oburationsmaterial analysieren, an dem Bakterien adhärieren [Karygianni et al., 2015]. Nur die Kombination einer adäquaten mikrobiologischen Analyse mit einem gut überdachten Probenentnahmeprotokoll kann die Zusammensetzung des Mikrobioms im Wurzelkanal klären.
Eine bestätigte Korrelation zwischen einer spezifischen endodontischen Infektionsflora und einer akuten oder chronischen apikalen Parodontitis kann zur Entwicklung neuer Wurzelkanal-Behandlungsstrategien führen. Dabei kommt der mikrobiologischen Analyse mittels moderner Techniken, die in zahlreichen medizinischen Fachdisziplinen mittlerweile Standard sind, eine Schlüsselrolle zu. Die Mikrobiom-Analyse könnte somit zu einer personalisierten Therapie speziell bei infizierten Nekrosen und sekundären Wurzelkanalinfektionen beitragen.
Mikrobiom-Analyse: next generation sequencing
Zu diesen modernen Techniken zählt das „next generation sequencing“ (NGS – Sequenzierung der nächsten Generation). In der Datenbank PubMed sind zum Thema „Mikrobiomanalyse oraler Nischen mittels Hochdurchsatz-Sequenziermethoden“ (NGS) aktuell 244 Arbeiten gelistet. Bei der Reduktion der Suche auf endodontische Infektionen bleiben lediglich 20 Studien übrig, die seit der Entwicklung der NGS zur Untersuchung der Mikrobiota endodontischer Infektionen im Zeitraum von zehn Jahren durchgeführt wurden. Dies zeigt, dass mittels NGS bisher nur sehr wenige Untersuchungen zur Charakterisierung endodontischer Infektionen im Vergleich zum supragingivalen und subgingivalen oralen Biofilm durchgeführt wurden.
Bei den NGS werden variable Regionen des 16S rRNA-Gens, das aus konservativen und neun variablen Regionen (V1–V9) besteht, sequenziert und anhand verschiedener Datenbanken bestimmten bakteriellen Taxa zugeordnet. Die Methoden erlauben die parallele Erfassung der Bakterien einer bestimmten Nische. Millionen von Sequenzabschnitten („Reads“) der amplifizierten variablen Regionen des 16S-rRNA-Gens werden gleichzeitig akquiriert und liegen dann in digitaler Form vor.
Sequenzen, die sich zu einem gewissen Prozentsatz ähneln (beispielsweise ab einer Übereinstimmung von 97 Prozent), werden über eine ausführliche Datenanalyse einer Spezies-Ebenen-„Operational Taxonomic Unit“ (OTU) zugeteilt [Braxter et al., 2005]. Eine solche OTU wird schließlich einer bestimmten Gattung oder einer einzelnen Spezies zugeordnet. Da alle Bakterien das 16S rRNA-Gen enthalten, werden durch die NGS auch Bakterien erfasst, die in den infizierten Wurzelkanälen nur selten vorkommen und durch die Kulturtechnik sowie die oben beschriebenen anderen molekularbiologischen Methoden nicht erkannt würden.
Die NGS erweist sich als Methode der Wahl
Mithilfe der Kulturtechnik und früherer molekularbiogischer Methoden (insbesondere der Klonierungstechnik) konnten bis zu 20 verschiedene Taxa aus primären beziehungsweise sekundären endodontischen Infektionen nachgewiesen werden [Anderson et al., 2013; Siqueira und Rôças, 2009; Manoil et al., 2020]. Ein Vergleich zwischen der Sanger-Sequenzierung nach Anwendung der Klonierungstechnik und der Pyrosequenzierung (NGS) gleicher endodontischer Proben zeigte, dass die NGS hoch signifikant mehr Phyla und Gattungen nachweisen [Li et al., 2010].
Betrachtet man die Ergebnisse der Mikrobiom-Analysen endodontischer Infektionen mittels NGS, die von Manoil et al. für primäre und sekundäre Infektionen in einem systematischen Review zusammengestellt wurden [Manoil et al., 2020], dann wird ersichtlich, dass die tatsächliche Diversität in diesen Infektionen bei durchschnittlich 400 OTUs lag. Diese hohe Zahl verschiedener Taxa gibt sowohl häufige als auch weniger häufige Spezies an. Das ist für die Beurteilung von endodontischen Misserfolgen beziehungsweise für die Entwicklung neuer Therapieverfahren von erheblicher Bedeutung, denn auch weniger häufige Spezies können eine Schlüsselrolle bei endodontischen Infektionen spielen. Zudem kann nicht ausgeschlossen werden, dass eine in geringer Häufigkeit nachgewiesene Art zu einem früheren oder späteren Zeitpunkt der Infektion eine wichtige Rolle bei der Pathogenität der Wurzelkanal-Mikrobiota spielt. Dies zeigt, dass die Mikrobiom-Analyse endodontischer Infektionen mittels NGS die Methode der Wahl ist, um die Ökologie der apikalen Parodontitis zu verstehen.
In früheren Studien konnten zwar auch mittels anderer Methoden immer wieder neue Arten aus infizierten Wurzelkanälen isoliert und identifiziert werden [Al-Ahmad et al., 2008; Schirrmeister et al., 2009; Anderson et al, 2012; Tennert et al, 2014; Siqueira und Rôças, 2009), eine Assoziation dieser Arten mit der klinischen Manifestation bleibt jedoch fraglich, da viele andere Arten, die die NGS in den letzten zehn Jahren nachwiesen, unabhängig von der Problematik der Probeentnahme nicht nachweisbar blieben [Manoil et al., 2020].
Im Unterschied dazu ist die Assoziation einer bestimmten Bakterien-Diversität mit symptomatischen oder nicht symptomatischen sekundären endodontischen Infektionen, wie dies mittels NGS von Anderson et al. [2013] gezeigt wurde, eher als evident zu betrachten. Die Autoren haben zum Beispiel erstmalig neue Phyla (TM7, Deinococcus-Thermus, Cyanobacteria, Chloroflexi, SR1 und OD1) nachgewiesen, die mithilfe früherer Methoden noch nicht in sekundären endodontischen Infektionen detektiert wurden. Auch Santos et al. konnten mittels der Mikrobiom-Analyse durch NGS unterschiedliche Mikrobiota-Diversitäten für akute (symptomatische) und chronische (asymptomatische) Infektionen nachweisen [Santos et al., 2011]. Gleichzeitig muss betont werden, dass alle bisherigen Mikrobiom-Analysen eine unspezifische, hoch diverse Mikrobiota mit unterschiedlichen Abundanzen der nachgewiesenen Taxa in den primären beziehungsweise sekundären Infektionen ergeben haben [Manoil et al., 2020].
Interessanterweise gibt es zu den Unterschieden zwischen primären und sekundären endodontischen Infektionen auch bei der Anwendung der Mikrobiom-Analyse durch NGS konträre Ergebnisse. Tzanetakis et al. berichteten von signifikanten Unterschieden der mikrobiellen Zusammensetzung zwischen primären und persistierenden endodontischen Infektionen [Tzanetakis et al., 2015]. Dagegen haben Keskin et al. und Hong et al. berichtet, dass die Mikrobiome beider Infektionstypen nicht signifikant unterschiedlich sind [Keskin et al., 2017; Hong et al., 2013].
Apikale und koronale Region: Die Unterschiede
Ein interessanter Befund, der nur durch die Mikrobiom-Analyse mittels NGS evident gezeigt werden konnte, ist der Unterschied der mikrobiellen Diversität zwischen der apikalen und der koronalen Region primärinfizierter Wurzelkanäle [Özok et al., 2012]. Die Autoren konnten durch eine Kryotechnik die infizierten Wurzelkanäle in unterschiedliche Segmente teilen, um die Mikrobiome dieser Segmente getrennt mittels NGS zu untersuchen.
Dadurch konnte gezeigt werden, dass die apikale Region diversere Mikrobiota enthält als die koronale Region. Das heißt, genau in dem klinisch schwer zu behandelnden Bereich befindet sich möglicherweise ein komplexes, mit den heutigen Aufbereitungs- und Spülsystemen schwer zu beeinflussendes Mikrobiom, das für die endodontischen Misserfolge mit verantwortlich ist.
NGS-Analysen bestätigen die Rolle bestimmter bekannter anaerober Firmicutes-Vertreter wie Pseudoramibacter alactolyticus, Dialisterpneumosintes oder Parvimonas micra in primären endodontischen Infektionen, aber lieferten gleichzeitig detailliertere Ergebnisse für die Rolle von Vertretern des Phylums Synergistetes wie den Gattungen Pyramidobacter und Fretibacterium in sekundären endodontischen Infektionen [Gomes et al., 2015; Özok et al., 2012; Bouillaguet et al., 2018; Manoil et al., 2020; Siqueira et al., 2016; Zandi et al., 2018]. Auch die häufig diskutierte Spezies Enterococcus faecalis erwies sich durch die NGS-Analyse eher als ein opportunistischer Infektionskeim, der durch die Behandlung primärer Infektionen in die Wurzelkanäle gelangt und Bestandteil sekundärer endodontischer Infektionen wird, ohne zu den Mikrobiota primärer Infektionen zu gehören, wie detailliert durch die Literaturanalyse bisheriger NGS-Studien von Manoil et al. gezeigt werden konnte [Manoil et al., 2020].
Probleme bei der Anwendung
Aber auch die neuen Analyseverfahren zur Mikrobiom-Analyse bei endodontischen Infektionen sind nicht immer fehlerfrei. Shin et al. haben die unterschiedlichen Techniken der NGS in ihrem Review miteinander verglichen und wiesen auf etliche Problematiken der Analysen hin [Shin et al., 2018].
So betonten die Autoren, dass in den bisherigen NGS-Studien zur Aufklärung endodontischer Infektionen unterschiedliche variable Regionen des 16S rRNA-Gens (insgesamt V1–V2, V3–V4), unterschiedliche Probenentnahmetechniken, kleine und auch unterschiedliche Probenzahlen (Patientenzahlen) aus sehr unterschiedlichen Populationen sowie unterschiedliche Strategien zur Sequenzanalyse und der Definition der OTUs verwendet wurden. Dies sind alles Faktoren, die zu konträren Ergebnissen geführt haben könnten.
Insbesondere die Probenentnahme kann eine große Rolle spielen. Die Verwendung von Paper Points würde wesentlich weniger Probenmaterial erfassen als die Verwendung von Mikropipetten oder die Extraktion der gesamten Zähne und die Pulverisierung der Wurzelkanäle nach Anwendung der Kryotechnik [Shin et al., 2018; Manoil et al., 2020]. Auch die Anwendung verschiedener DNA-Extraktionsprotokolle kann zu unterschiedlichen DNA-Ausbeuten der verschiedenen Taxa führen und damit die Ergebnisse der Mikrobiom-Analyse beeinflussen. Manoil et al. [2020] betonten auch die Bedeutung der gleichzeitigen Durchführung von Negativkontrollen, die eine Kontamination der entnommenen Proben, insbesondere bei der Verwendung von kompletten Wurzelkanälen nach Zahnextraktion, ausschließen sollen.
Um Variationen durch die angewandte PCR, Sequenziertechnik und Datenverarbeitung auszuschließen, sollte eine sogenannte Mock Community mit einer definierten Artenzusammensetzung als interner Standard verwendet werden [Yeh et al., 2018]. In keiner der bisher durchgeführten Mikrobiom-Analysen zur Untersuchung endodontischer Infektionen durch NGS wurde eine solche interne Kontrolle verwendet. Fehler bei den erhaltenen Ergebnissen können dadurch nicht ausgeschlossen werden. Dies liegt auch daran, dass es nicht genug Erfahrung in der Mikrobiom-Analyse infizierter Wurzelkanäle gibt, da in diesem Bereich bisher nur sehr wenige Studien durchgeführt wurden.
Zusätzlich zur Untersuchung der Mikrobiota schlagen Zehnder und Belibasakis [2015] vor, auch eine parallele Analyse von Immunmediatoren in den entnommenen mikrobiologischen Proben durchzuführen, um die Immunantwort des Wirts und damit das Krankheitsbild besser verstehen und mit den nachgewiesenen Mikroorganismen assoziieren zu können.
Fazit und Ausblick
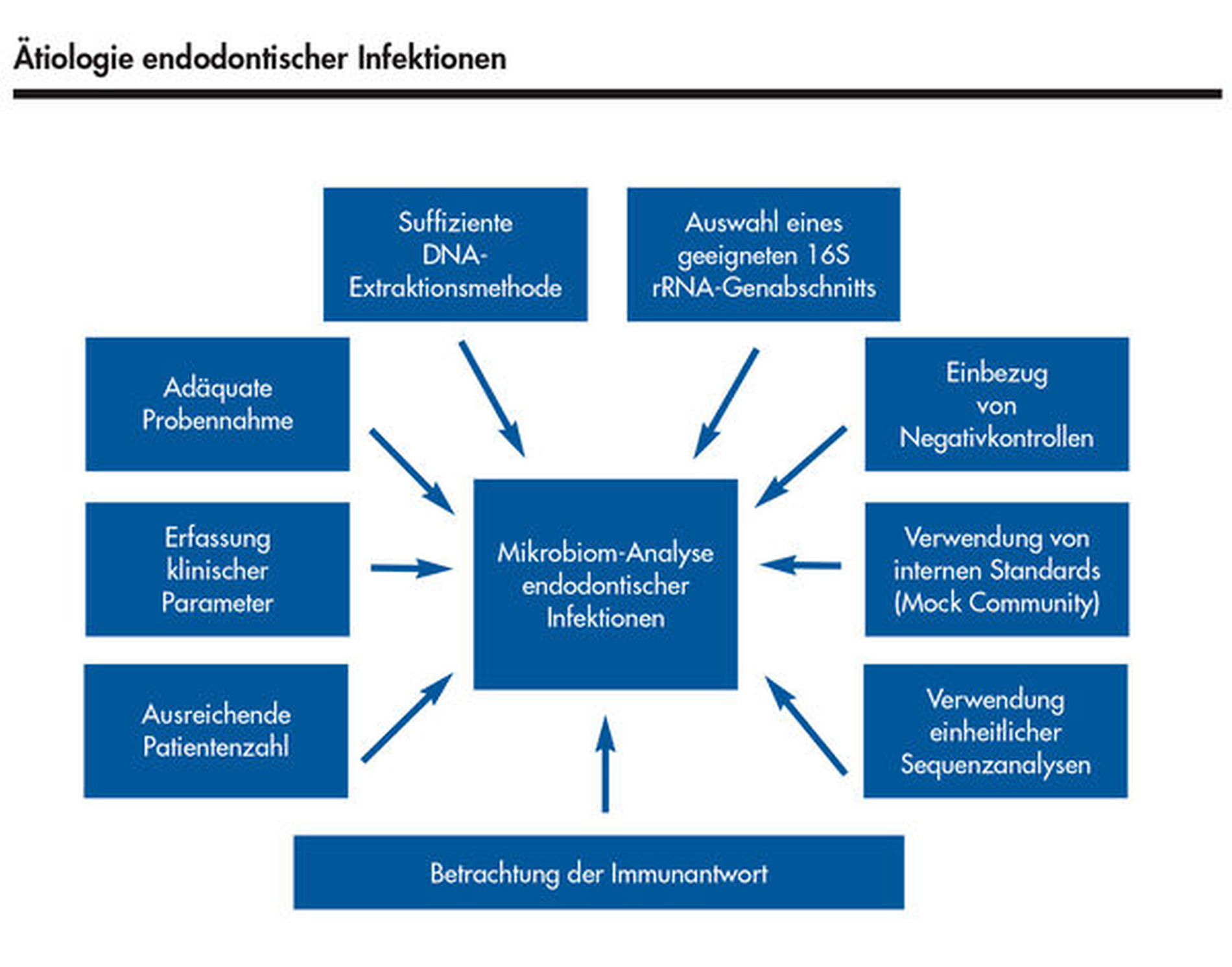
Es ist mittlerweile erwiesen, dass eine Mikrobiom-Analyse durch NGS notwendig ist, um die Ätiologie primärer und speziell sekundärer endodontischer Infektionen besser zu verstehen und damit zukünftig auch Zähne erhalten zu können, die heute aufgrund einer refraktären Parodontitis apicalis extrahiert werden müssen. Dazu müssen mehr klinische Studien durchgeführt werden, bei denen auf folgende Prämissen geachtet werden sollte:
eine ausreichende Patientenzahl,
die Erfassung der notwendigen klinischen Parameter der Patienten,
eine adäquate Probenentnahme zur Erfassung von möglichst allen beteiligten Mikroorganismen,
die Auswahl einer suffizienten DNA-Extraktionsmethode, die Auswahl von möglichst langen Abschnitten von mehreren variablen Regionen des 16S rRNA-Gens, um eine Erfassung der Mikrobiota auf Speziesebene zu ermöglichen,
den Einbezug von Negativkontrollen zur Vermeidung von Kontamination mit der oralen Mikrobiota,
das Heranziehen eines internen Standards (Mock Community), um Variationen durch die angewandte PCR und Sequenziertechnik sowie Datenverarbeitung auszuschließen,
die Verwendung von einheitlichen Sequenzanalysen und spezifischen statistischen Methoden sowie den Vergleich der erhaltenen Sequenzen mit möglichst verschiedenen internationalen Sequenzdatenbanken.
Auf der Basis dieser grundlegenden Studien könnten dem Zahnarzt künftig Therapiemaßnahmen an die Hand gegeben werden, mit denen es gelingt, auch endodontische Problemzähne zu erhalten. So wäre durchaus denkbar, in solchen Fällen auf der Basis von definierten Probenentnahmeverfahren aus dem Wurzelkanal spezielle antibakterielle Verfahren einzusetzen, die über die Standardtherapie hinausgehen.
Eine zusätzliche Betrachtung der Immunantwort des Patienten würde die Interpretation der Ergebnisse einer Mikrobiom-Analyse bei endodontischen Infektionen und die Vorhersagbarkeit eines Therapieerfolgs verbessern. Abbildung 2 zeigt schematisch die notwendigen Voraussetzungen für eine erfolgreiche Mikrobiom-Analyse endodontischer Infektionen.
Literaturliste
1. Al-Ahmad A, Pelz K, Schirrmeister JF, Hellwig E, Pukall R. Characterization of the first oral vagococcus isolate from a root-filled tooth with periradicular lesions. Curr Microbiol. 2008; 57:235-238.
2. Anderson AC, Hellwig E, Vespermann R, Wittmer A, Schmid M, Karygianni L, Al-Ahmad A. Comprehensive analysis of secondary dental root canal infections: a combination of culture and culture-independent approaches reveals new insights. PLoS One. 2012; 7(11):e49576.
3. Anderson AC, Al-Ahmad A, Elamin F, Jonas D, Mirghani Y, Schilhabel M, Karygianni L, Hellwig E, Rehman A. Comparison of the bacterial composition and structure in symptomatic and asymptomatic endodontic infections associated with root-filled teeth using pyrosequencing. PLoS One 2013; 8:e84960.
4. Blaxter M, Mann J, Chapman T, Thomas F, Whitton C, Floyd R, Abebe E: Defining operational taxonomic units using DNA barcode data. Philos Trans R Soc Lond B Biol Sci. 2005; 360:1935-1943.
5. Bouillaguet S, Manoil D, Girard M, Louis J, Gaia N, Leo S, Schrenzel J, Lazarevic V. Root Microbiota in Primary and Secondary Apical Periodontitis. Front Microbiol. 2018; 9: 2374.
6. Dewhirst FE, Chen T, Izard J, Paster BJ, Tanner AC, Yu WH, Lakshmanan A, Wade WG. The human oral microbiome. J Bacteriol. 2010; 192:5002-5017.
7. Gomes BP, Berber VB, Kokaras AS, Chen T, Paster BJ. Microbiomes of Endodontic-Periodontal Lesions before and after Chemomechanical Preparation. J Endod. 2015; 41:1975-84.
8. Hong B-Y, Lee T-K, Lim S-M, Chang SW, Park J, Han SH, Zhu Q, Safavi KE, Fouad AF, Kum KY. Microbial analysis in primary and persistent endodontic infections by using pyrosequencing. J Endod. 2013; 39:1136-1140.
9. Iriboz E, Öztürk BA, Kolukırık M, I Karacan I, H Sazak Öveçoğlu HS. Detection of the unknown components of the oral microflora of teeth with periapical radiolucencies in a Turkish population using next-generation sequencing techniques. Int Endod J. 2018; 51:1349-1357.
10. Kakehashi S, Stanley H, Fitzgerald R. The effects of surgical exposures of dental pulps in germ-free and conventional laboratory rats. Oral Surg Oral Med Oral Pathol. 1965; 20:340–349.
11. Karygianni L, Anderson AC, Tennert C, Kollmar K, Altenburger MJ, Hellwig E, Al-Ahmad A. Supplementary sampling of obturation materials enhances microbial analysis of endodontic treatment failures: a proof of principle study. Clin Oral Investig. 2015; 19:319-27.
12. Keskin C, Demiryürek EÖ, Onuk EE. Pyrosequencing Analysis of Cryogenically Ground Samples from Primary and Secondary/Persistent Endodontic Infections. J Endod. 2017; 43:1309-1316.
13. Kirkevang LL, Vaeth M, Hörsted-Bindslev P, Bahrami G, Wenzel A. Risk factors for developing apical periodontitis in a general population. Int Endod J. 2007; 40:290-299.
14. Li L, Hsiao WW, Nandakumar R, Barbuto SM, Mongodin EF, Paster BJ, Fraser-Liggett CM, Fouad AF. Analyzing endodontic infections by deep coverage pyrosequencing. J Dent Res. 2010; 89:980-984.
15. Manoil D, Al-Manei K, Belibasakis GN. A Systematic Review of the Root Canal Microbiota Associated with Apical Periodontitis: Lessons from Next-Generation Sequencing. Proteomics Clin Appl. 2020 May; 14(3):e1900060.
16. Moreno JO, Alves FR, Goncalves LS, et al. Periradicular status and quality of root canal fillings and coronal restorations in an urban Colombian population. J Endod. 2013; 39:600-604.
17. Nair PNR, Henry S, Cano V, Vera J. Microbial status of apical root canal system of human mandibular first molars with primary apical periodontitis after “one-visit” endodontic treatment. Oral Surg. Oral Med. Oral Pathol. Oral Radiol. Endodontology 2005; 99: 231–252.
18. Özok AR, Persoon IF, Huse SM, Keijser BJF, Wesselink PR, Crielaard W, Zaura E. Ecology of the microbiome of the infected root canal system: a comparison between apical and coronal root segments. Int Endod J. 2012; 45:530–541.
19. Paster BJ, Boches SK, Galvin JL, Ericson RE, Lau CN, Levanos VA, Sahasrabudhe A, Dewhirst FE. Bacterial diversity in human subgingival plaque. J Bacteriol. 2001; 183:3770-3783.
20. Ricucci D, Siqueira JF Jr. Biofilms and apical periodontitis: study of prevalence and association with clinical and histopathologic findings. J Endod. 2010; 36:1277-1288.
21. Saito D, de Toledo Leonardo R, Rodrigues JLM, Tsai SM, Höfling JF, Gonçalves RB. Identification of bacteria in endodontic infections by sequence analysis of 16S rDNA clone libraries. J Med Microbiol. 2006; 55:101-107.
22. Saito D, Marsh TL, de Souza Cannavan F, Höfling JF, Gonçalves RB. Assessment of intraradicular bacterial composition by terminal restriction fragment length polymorphism analysis. Oral Microbiol Immunol. 2009 Oct; 24(5):369-76.
23. Santos AL, Siqueira JF Jr, Rôças IN, Jesus EC, Rosado AS, Tiedje JM.Comparing the bacterial diversity of acute and chronic dental root canal infections. PLoS One. 2011; 6(11):e28088.
24. Sakamoto M, Siqueira JF Jr, Rôças IN, Benno Y. Molecular analysis of the root canal microbiota associated with endodontic treatment failures. Oral Microbiol Immunol. 2008 Aug; 23(4):275-81.
25. Sathorn C, Parashos P, Messer HH. How useful is root canal culturing in predicting treatment outcome? J of Endod. 2007; 33:220–225.
26. Schirrmeister JF, Liebenow AL, Braun G, Wittmer A, Hellwig E, Al-Ahmad A. Detection and eradication of microorganisms in root-filled teeth associated with periradicular lesions: an in vivo study. J Endod. 2007; 33:536-540.
27. Schirrmeister JF, Liebenow AL, Pelz K, Wittmer A, Serr A, Hellwig E, Al-Ahmad A. New bacterial compositions in root-filled teeth with periradicular lesions. J Endod. 2009; 35:169-174.
28. Shin JM, Luo T, Lee KH, Guerreiro D, Botero TM, McDonald NJ, Rickard AH. Deciphering Endodontic Microbial Communities by Next-generation Sequencing. J Endod. 2018; 44:1080-1087.
29. Siqueira JF Jr, Rôças IN. Diversity of endodontic microbiota revisited. J Dent Res. 2009; 88:969-981.
30. Siqueira JFJ, Rocas IN, Rosado AS. Investigation of bacterial communities associated with asymptomatic and symptomatic endodontic infections by denaturing gradient gel electrophoresis fingerprinting approach. Oral Microbiol Immunol. 2004; 19:363-370.
31. Siqueira JF Jr, Rôças IN. Uncultivated phylotypes and newly named species associated with primary and persistent endodontic infections. J Clin Microbiol. 2005 Jul; 43(7):3314-9.
32. Siqueira JF Jr., Antunes HS, Rocas IN, Rachid CT, Alves FR. Microbiome in the Apical Root Canal System of Teeth with Post-Treatment Apical Periodontitis. PLoS One. 2016; 11: e0162887.
33. Tennert C, Fuhrmann M, Wittmer A, Karygianni L, Altenburger MJ, Pelz K, Hellwig E, Al-Ahmad A. New bacterial composition in primary and persistent/secondary endodontic infections with respect to clinical and radiographic findings. J Endod. 2014; 40:670-677.
34. Tzanetakis GN, Azcarate-Peril AM, Zachaki S, Panos Panopoulos P, Kontakiotis EG, Phoebus N. Madianos PN, Divaris K. Comparison of Bacterial Community Composition of Primary and Persistent Endodontic Infections Using Pyrosequencing. J. Endod. 2015; 41:1226-1233.
35. Wade WG. The oral microbiome in health and disease. Pharmacol Res. 2013; 69:137-143.
36. Yeh YC, Needham DM, Sieradzki ET, Fuhrman JA. Taxon Disappearance from Microbiome Analysis Reinforces the Value of Mock Communities as a Standard in Every Sequencing Run. mSystems. 2018; 3:e00023-18.
37. Zandi H, Kristoffersen AK, Orstavik D, Rocas IN, Siqueira JF Jr, Enersen M. Microbial Analysis of Endodontic Infections in Root-filled Teeth with Apical Periodontitis before and after Irrigation Using Pyrosequencing. J Endod. 2018; 44: 372-378.
38. Zehnder M, Belibasakis GN. On the dynamics of root canal infections - whatwe understand and what we don’t. Virulence. 2015; 6:3, 216-222.






